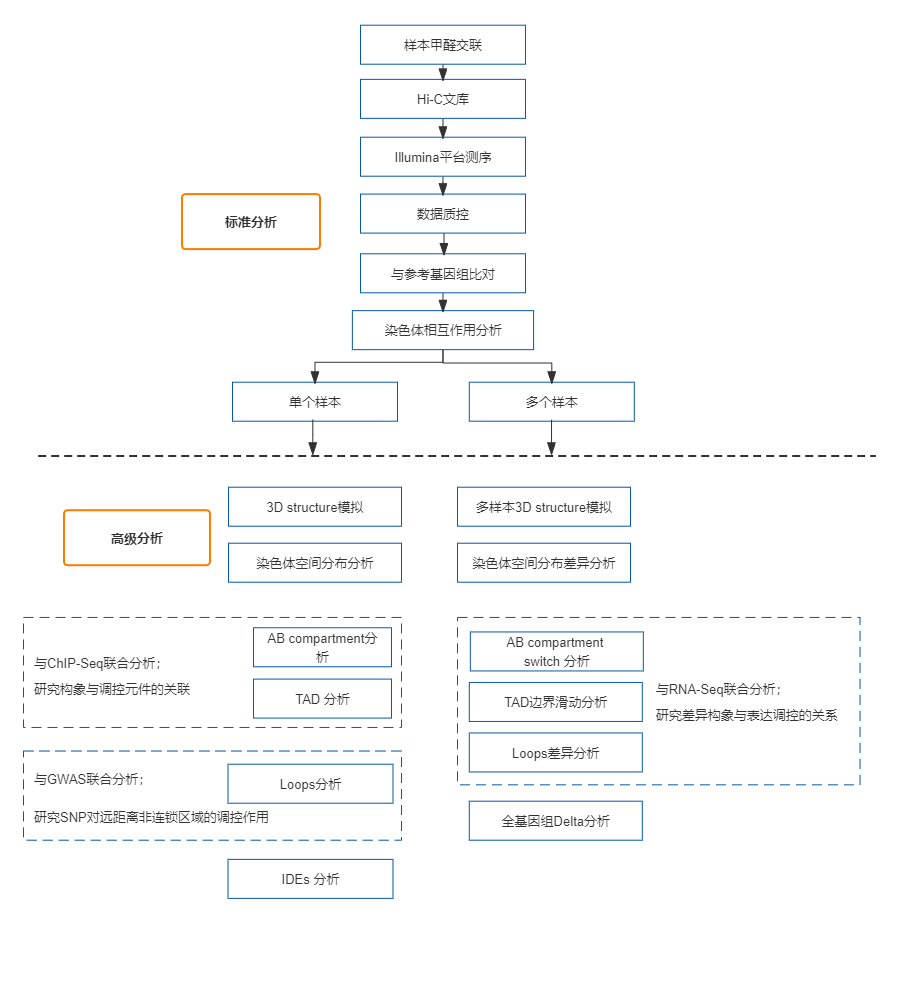
Hi-C技术源于染色体构象捕获(Chromosome Conformation Capture—3C)技术,以整个细胞核为研究对象,利用高通量测序技术,结合生物信息学方法,研究全基因组范围内整个染色质DNA在空间位置上的关系;通过对染色质内全部DNA相互作用模式进行捕获,获得高分辨率的染色质三维结构信息,并与ATAC-seq、ChIP-seq、基因组、转录组等数据联合分析,从基因调控网络和表观遗传网络来阐述生物体性状形成的相关机制。
建库技术
有效数据比例可达90%以上,1万细胞即可建库
物种经验
成功实现对人、鼠、猴、猪、牛、鸡、家蚕等物种稳定建库
文章经验
合作文章发表于Nature、Cell等期刊,累积影响因子200+
分析软件
自主研发HiC-Pro、HiTC等信息分析软件
分析内容
实现与ATAC-seq、ChlP-seq、WGBS、RNA-seq、三代重测序等多种组学联合分析
染色质结构解析
基因表达调控
结构变异检测
疾病机制研究
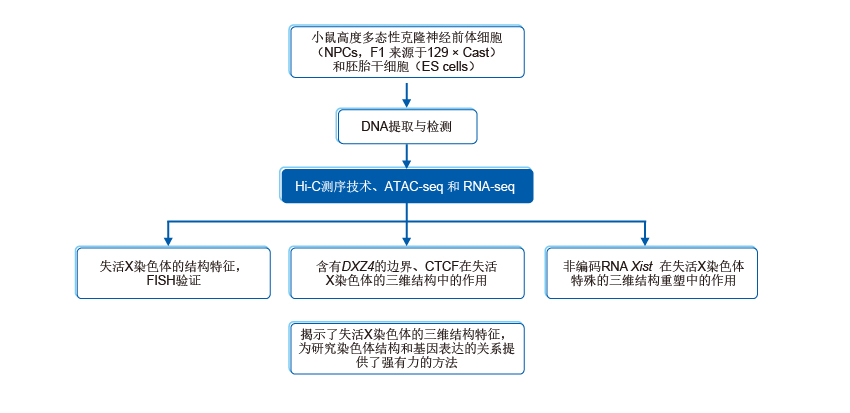
小鼠失活X染色体结构特征
Structural organization of the inactive X chromosome in the mouse
期刊:Nature 发表时间:2016 影响因子:38.138 合作单位:法国居里研究所
失活X染色体的结构特征
为了研究失活X染色体的结构,选取来源于高度多态性F1代小鼠胚胎干细胞的神经前体细胞做等位基因特异的Hi-C分析。首先,在胚胎干细胞中做Hi-C分析,ES细胞中还没有发生X染色体沉默;发现活跃的X染色体含有明显的活跃、 失活区室化(compartment A/B),也没有拓扑结构域结构(TAD)(图1)。
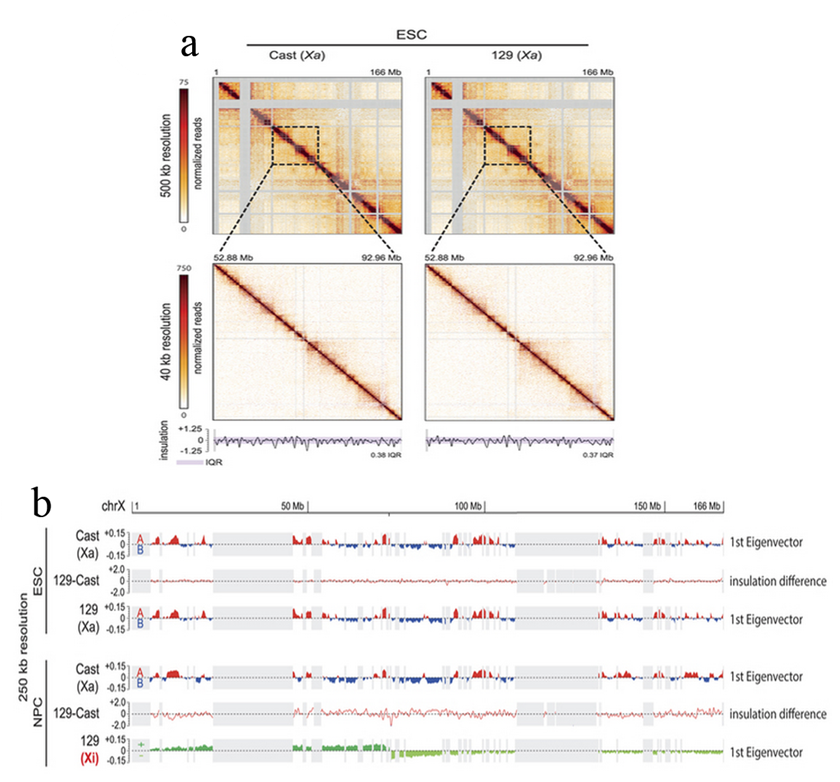
图1 胚胎干细胞染色质三维结构特征
高度多态性F1代小鼠胚胎干细胞的神经前体细胞中(NPCs),在常染色质和未失活的X染色体中具有类似的A/B compartment和TAD结构;但是在失活的X染色体中没有A/B compartment(图1),而是由含有DXZ4微卫星的200kb铰链区域分割成两个大的相互作用区域;此外,在失活的X染色体中也没有拓扑结构域(TADs)(图2)。
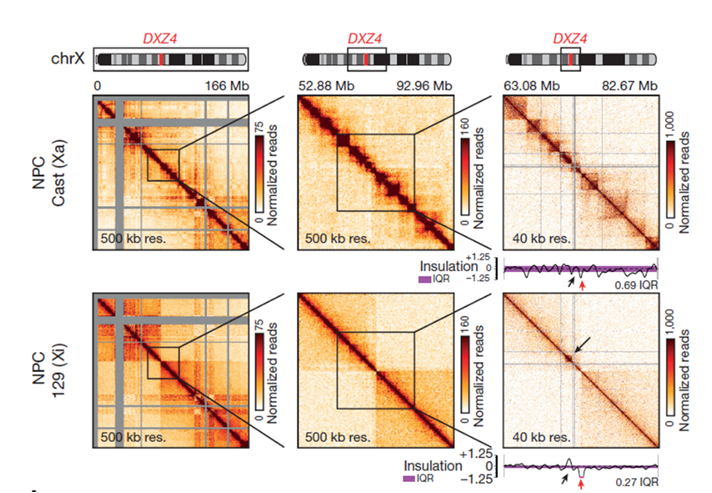
图2 失活的X染色体结构特征
含有DXZ4的边界、CTCF在失活X染色体的三维结构中的作用
为了研究沉默X染色体中不寻常的两个结构域的重要作用,我们删除了小鼠胚胎干细胞中来源于129亲本等位基因中含有DXZ4位点的200kb边界区域。胚胎干细胞经分化后,分离出许多含有129 亲本X染色体沉默的NPC克隆(ΔFT/D9B2克隆),Hi-C分析表明失活X染色体结构重塑导致了两个大的结构域发生融合(图3a)。ATAC-seq和RNA-seq表明,在以上分化后的NPC克隆中,87个兼性逃离沉默的基因中有66个失去了转录和染色质亲和特性(图3b)。比较D9B2和野生型NPCS 中沉默的X染色体发现,二者在沉默基因的避免、染色质亲和性缺失和TAD信号减弱有很高的相关性(图3c)。在删除含有DXZ4边界的失活X染色体中,特殊的远距离互作发生了缺失(图3d)。在野生型克隆中用ATAC-seq在沉默的X染色体中检测到的224个peak中,139个在D9B2中是缺失的(图3e);这些缺失位点富集在启动子附近含有CTCF结合位点的区域(图3f,g),这些CTCF更接近于“逃离沉默”的基因转录起始位点(TSSs),再次证明了启动子附近的CTCF在“逃离沉默”的基因调控中发挥着重要作用(图3h)。
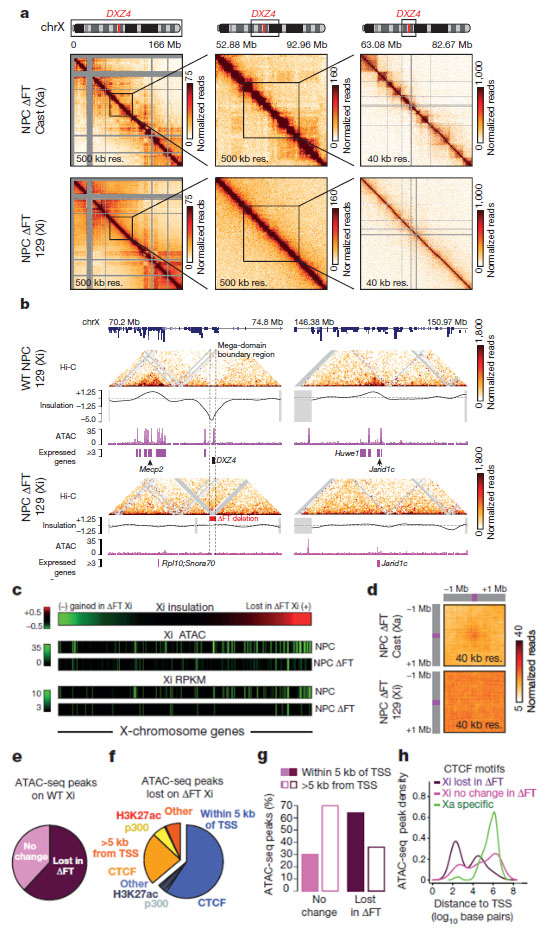
图3 含有DXZ4的边界、CTCF在失活X染色体的三维结构中的作用
Xist在失活X染色体特殊的三维结构重塑中的作用
在未分化的雄性小鼠ES细胞中诱导Xist表达,Hi-C分析发现诱导Xist 48小时导致X染色体结构发生明显变化(图 4a-b);但是在不含A重复区域的Xist突变样品中,基因不发生沉默,染色体三维结构没有变化。野生型Xist诱导不会导致TAD结构的显著改变,但是会导致染色体相互作用频率的增加(图4d), Mb级别的结构域分割边界用RNA/DNA FISH验证是正确的(图4c)。
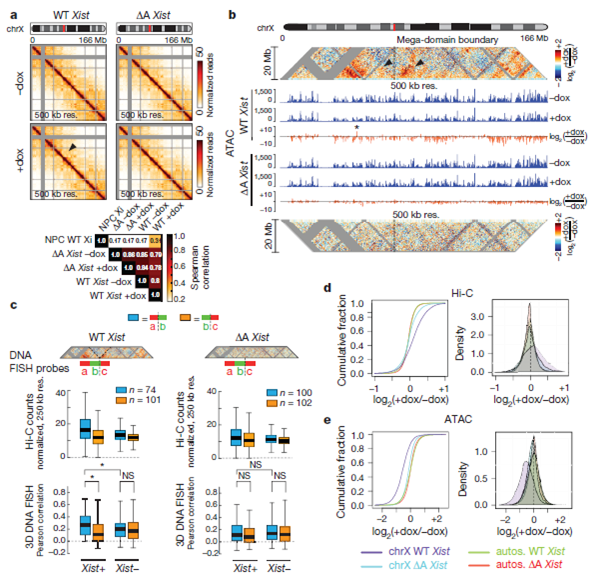
图4 Xist在失活X染色体特殊的三维结构重塑中的作用
本研究揭示了失活X染色体的三维结构特征,为研究染色体结构和基因表达的关系提供了强有力的方法,将帮助科学家们更多地了解活细胞中基因组的运作情况;同时有助于开发新的方法,对抗女性X染色体相关的疾病,促进相关疾病的研究与治疗。
Giorgetti L, Lajoie B R, Carter A C, et al. Structural organization of the inactive X chromosome in the mouse[J]. Nature, 2016, 535(7613): 575-9.