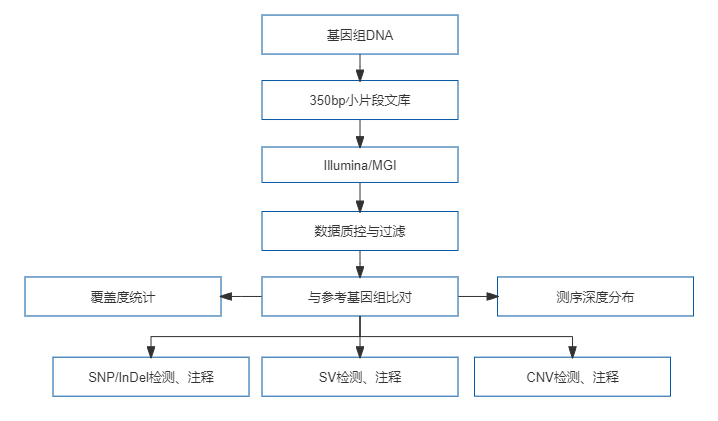
变异检测是指通过高通量测序技术对某一物种个体或群体的基因组进行测序及差异分析,获得大量的遗传变异信息,如单核苷酸多态性(SNP)、插入缺失(InDel)、结构变异(SV)、拷贝数变异(CNV)等,可用于开发分子标记建立遗传多态性数据库,为后续揭示进化关系、挖掘功能基因等奠定数据基础。
籼稻辐照诱导突变体的全基因组变异分析
Genome-wide analysis of radiation-induced mutations in rice (Oryza sativa L. ssp. indica)
期 刊: Molecular Biosystems 发表时间: 2014.10 发表单位: 福建农林大学 影响因子: 3.210
物理辐照已经被成功用于水稻种植资源创新。然而,通过辐照诱发突变的分子机制仍不清楚。在本研究中,研究者通过全基因组重测序技术揭示辐照诱变处理之后水稻基因组的变异情况。
材料:水稻9311经γ射线诱变处理得到红色种皮和大米有益成分含量增加的突变材料M1,经过6代自交得到纯系材料Red-1
建库:叶片组织基因组总DNA,构建小片段文库
测序:Solexa 双末端测序,20X
分析:SNP检测、Indel检测、SV检测、基因功能富集分析
Red-1材料的基因密度在200kb/gene,全基因组重测序检测到381,403个SNPs,50,116个1-5bp的Indels,1,279个CNVs和10,026个PAVs,点突变是主要的突变类型。
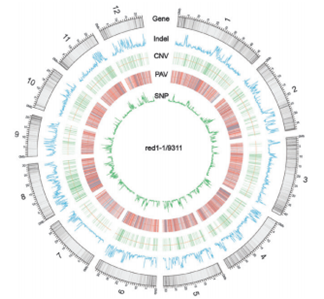
图1 变异在水稻基因组中的分布
检测到的SNP分布于8,724个基因,Indels覆盖了6,271个基因,SV覆盖了5,016个基因。变异的分布涵盖了基因的不同区域,存在一定染色体偏好性,主要集中在第1、2、3、4和8号染色体。基因功能主要涉及激酶结构域、植物细胞色素_p450结构域、亮氨酸重复结构域等。
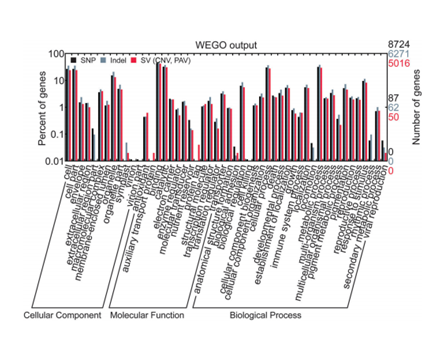
图2 SNPs、Indels和SVs三种变异类型相关的基因WEGO注释
KEGG功能富集分析发现,参与细胞组分、结合功能以及催化活性、代谢过程相关相关基因易受γ射线辐照影响,筛选到8个基因可能涉及突变体有益成分的生物合成途径及色素调控。
全基因组变异检测为研究辐射引起水稻Red-1中种皮颜色和有益成分变异的分子机制提供新的研究思路。
Cheng Z, Lin J, Lin T, et al. Genome-wide analysis of radiation-induced mutations in rice (Oryza sativa L. ssp. indica)[J]. Molecular Biosystems, 2014, 10(4): 795-805.