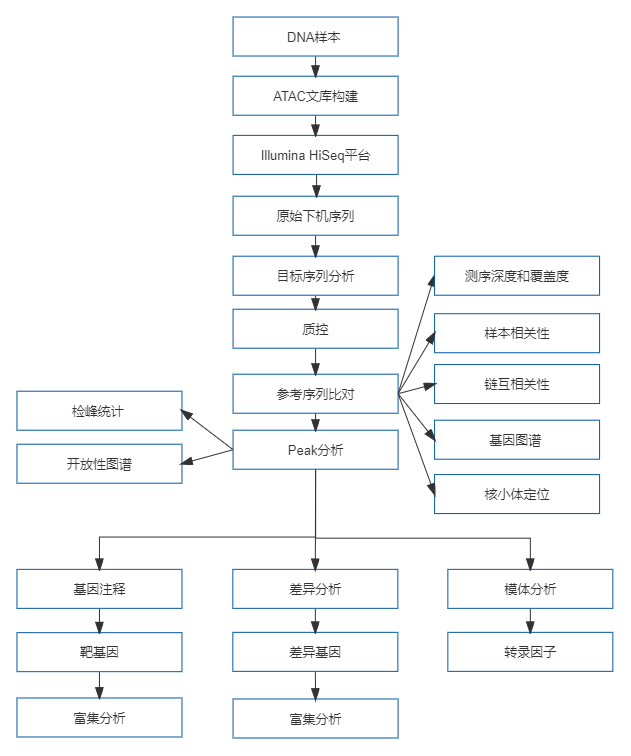
ATAC-seq(Assay for Transposase-Accessible Chromatin with high-throughput sequencing)是利用DNA转座酶(Tn5)切割开放的DNA区域并结合二代高通量测序技术来研究染色质开放性的新技术。与传统研究染色质开放性的MNase-seq和DNase-seq相比,ATAC-seq的可重复性更强,实验步骤和操作简单、省时,只需要很少量的细胞/组织就能鉴定基因组中的全部开放区域,是目前研究染色质可接近性的方法。
商业化项目经验
合作项目200+
样品建库经验
人、鼠在内的多物种细胞和组织建库
生信分析
标准分析、多组学联合分析、定制化分析
项目周期
40天完成建库测序及标准分析
期刊:Nature 发表时间:2022.2 影响因子:49.962 合作单位:中科院遗传发育所、中科院微生物所
破坏作物的感病(S)基因是赋予作物抗病能力的一种具有前景的育种策略。但破坏S基因的行为是一把双刃剑:在作物抗病性提升的同时,往往是以牺牲产量为代价的,这极大地限制了其在植物抗病育种中的应用。
近日中科院遗传发育所高彩霞团队、中科院微生物所邱金龙团队和中科院遗传发育所肖军团队合作发表在Nature(IF=49.962)的研究[1]中,发现感病基因MLO突变的Tamlo-R32突变体不仅被赋予强大的白粉病抗性,同时也保留了小麦的生长与产量的优势。作者通过cut&tag、ATAC-seq、RNA-seq与基因组编辑等在内的多组学技术手段,成功对小麦白粉病抗性和产量兼得的遗传改良途径进行剖析。
安诺优达有幸为本研究提供了ATAC-seq、4C测序和转录组建库测序服务,为挖掘新型突变体抗病高产的分子机制提供助力!带着满满的好奇心,下面就和小编一起来学习如何利用高通量测序技术,探索作物遗传改良的奥秘吧~

1、全基因组重测序:Novaseq PE150 测序深度50X
2、RNA-seq、4C-seq、ATAC-seq、CUT & Tag:NovaSeq PE150
1、Tamlo-R32抗白粉病,且无生长缺陷
作者在使用TALEN 载体产生的小麦mlo突变体中,成功鉴定了Tamlo-R32 突变体。该突变体表现出对白粉病强大的抗性,且株高和谷物产量方面没有表现出生长缺陷。
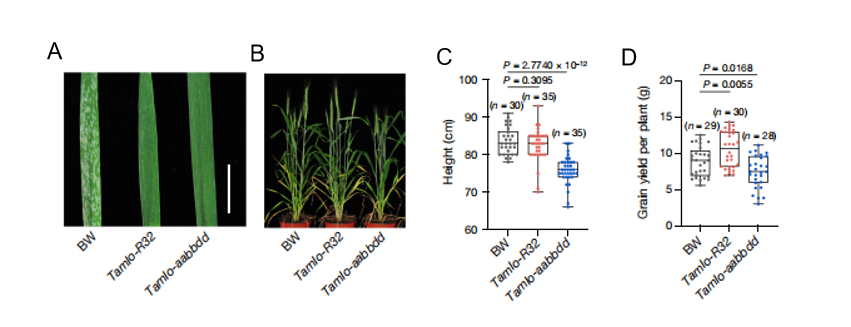
图1 Tamlo-R32抗白粉病且无生长缺陷
2、Tamlo-R32在TaMLO-B1附近产生304Kb大片段缺失
作者接下来对Tamlo-R32抗性相关的突变进行探究。Tamlo-R32 突变体中共有3个小麦MLO1基因,作者通过基因特异性引物扩增和基因杂交发现Tamlo-R32 中的抗性等位基因与 TaMLO-B1 基因座紧密相关。全基因组测序表明其染色体 4B 上发现了一个 304 Kb大片段缺失。缺失的右边界位于 TaMLO-B1 的第二个外显子,左边界终止于 MLO 基因(以下称为 TaMLOX)。
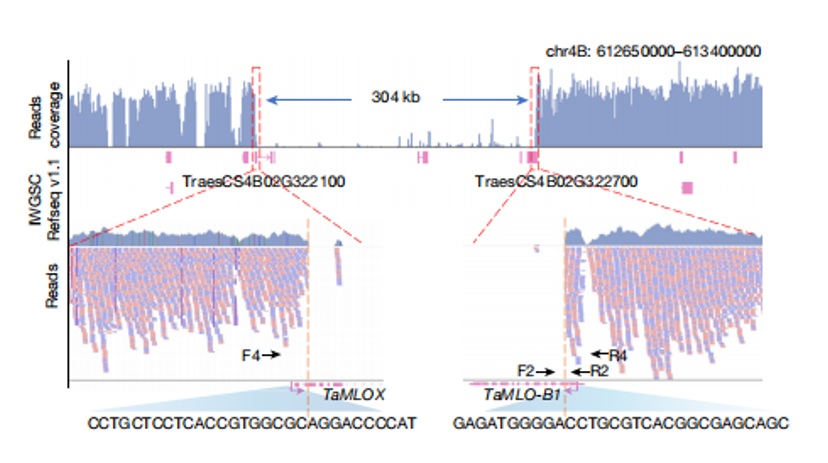
图2 Tamlo-R32在TaMLO-B1附近产生304Kb大片段缺失
3、挽救Tamlo-R32生长缺陷的TaTMT3B表达调控机制
为了研究Tamlo-R32中大片段缺失的影响,作者通过RNA-seq测序发现缺失上游的TaTMT3B基因相对于野生型 Bobwhite在 Tamlo-R32 中显著上调,进而导致TaTMT3 的总水平增加。
通过cut&tag和ATAC-seq检查TaTMT3B-MLO-B1 区域中的组蛋白修饰和染色质可及性,并辅以染色体构象捕获测序 (3C-seq) 和环状 3C-seq (4C-seq)技术,发现野生型 中 TaTMT3B 被 H3K27me3 (参与形成导致基因沉默的长程染色质环)标记的启动子与远端基因座成环,而Tamlo-R32中大片段的缺失消除了 TaTMT3B 启动子和缺失区域(a-c 环)之间的染色质环,表明TaTMT3B 的转录受到抑制性组蛋白标记 H3K27me3 和野生型 Bobwhite 中抑制性染色质环的存在的抑制,而 Tamlo-R32 中的片段缺失消除了表观遗传抑制,并在 TaTMT3B 周围创造了一个致其上调的染色质环境。
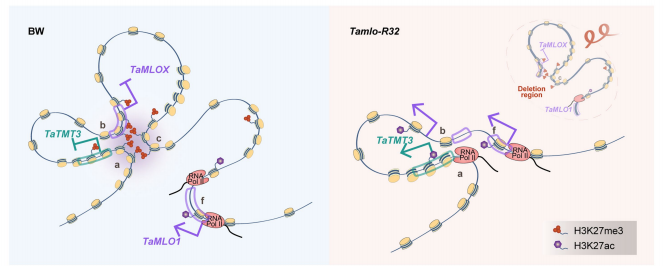
图3 TaTMT3B表达调控示意图
4、利用多重基因组编辑快速获得抗白粉病小麦新种质
基于以上研究结果,作者利用CRISPR-Cas9技术来开发含有 Tamlo-R32 等位基因的新小麦种质。使用仅具有两个 sgRNA 的 CRISPR–Cas9 DNA 或 RNP 将 Tamlo-R32 等位基因快速引入优质小麦品种,最终编辑四个优良冬小麦品种中的 MLO基因座。这些结果证明了相比于基因渗入,基因组编辑是将 Tamlo-R32 等位基因快速引入优质小麦品种的有力策略。
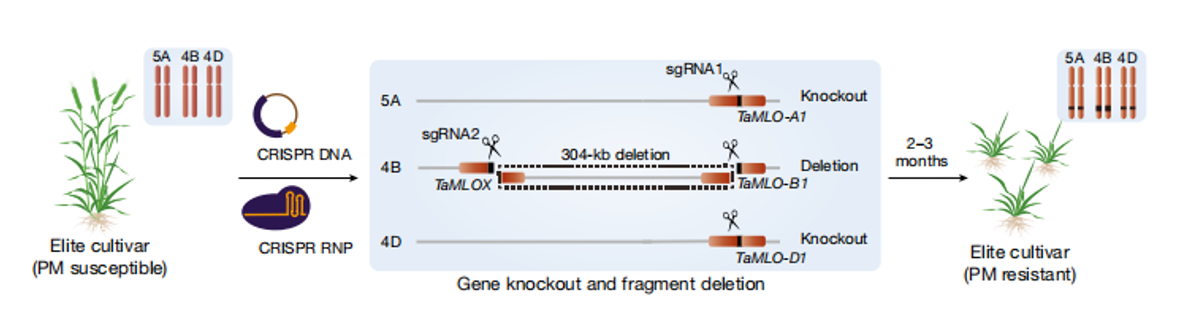
图4 使用CRISPR–Cas9 技术将 Tamlo-R32 等位基因快速引入优质小麦品种示意图
感病基因MLO的突变使病原微生物侵染能力下降,植株表现出抗病表型,但往往其生长和产量均受到负面影响。与其他突变体不同的是,Tamlo-R32突变体内发现一个304Kb染色体片段的缺失。作者通过染色体三维空间图的绘制与转录组分析,发现其染色质三维空间结构发生重排,表观的遗传变化导致上游编码液泡膜单糖转运基因TaTMT3显著上调,这恰好恢复了Tamlo-R32材料的生长和产量。该团队通过杂交和多代回交,将MLO突变和染色体缺失片段一起导入优良小麦品种中,获得了抗白粉病且高产品系的快速创新。
安诺优达在表观方面拥有多样化的产品类型,利用Hi-C、WGBS、ATAC-seq、ChIP-seq等技术助力您在染色体结构解析、基因表达调控、疾病机制、结构变异检测等方面的研究。安诺优达提供表观基因组、转录组等多组学的测序分析服务,针对性的设计项目执行方案,同时安诺云平台搭载多组学联合分析平台,提供100+在线小工具,助力您的深度个性化挖掘和结果展示!
[1] Li S, Lin D, Zhang Y, Deng M, Chen Y, Lv B, Li B, Lei Y, Wang Y, Zhao L, Liang Y, Liu J, Chen K, Liu Z, Xiao J, Qiu JL, Gao C. Genome-edited powdery mildew resistance in wheat without growth penalties. Nature. 2022 Feb 9.