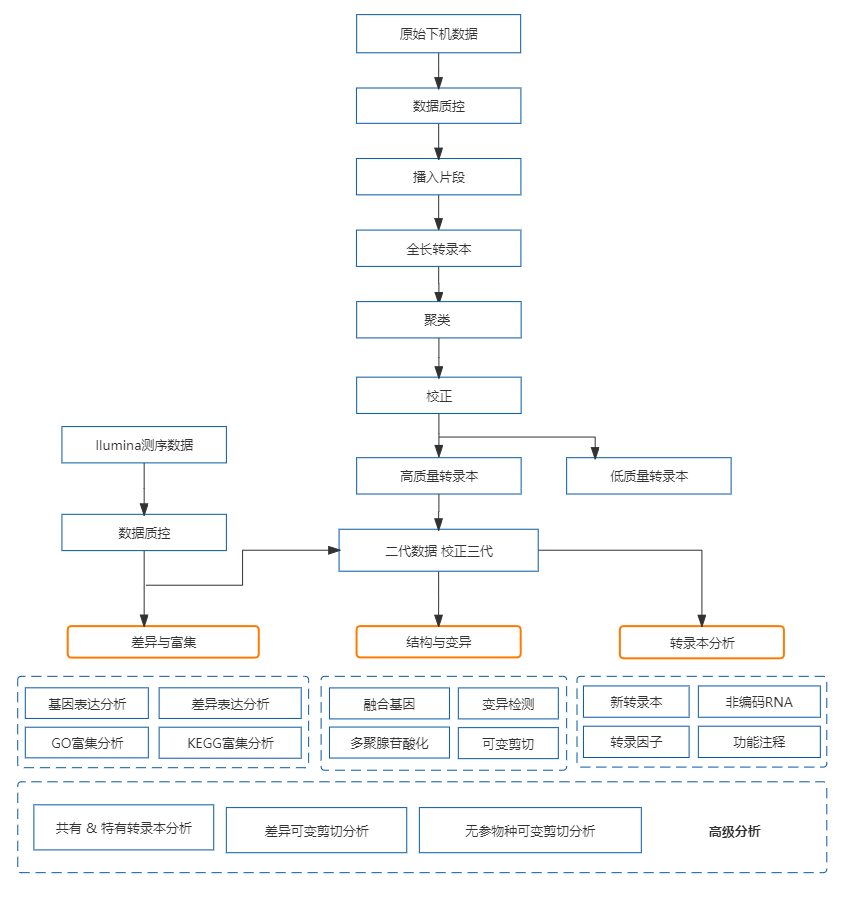
全长转录组测序(Iso-seq)是指利用三代测序技术对生物体内完整的RNA分子(包括5'末端到3'poly A尾的全长序列)进行测序,RNA分子无需打断,测序可直接获得完整序列。通过全长转录组测序能够更准确地得到转录本信息,可鉴定更多可变剪接位点、新基因和新的异构体、融合基因等。
揭示物种的转录组复杂性
辅助基因组注释
探究不同处理条件下的转录本差异
不同时空、组织的转录本结构、功能特异性
通过可变剪切、融合基因等探究疾病、肿瘤的发生机制
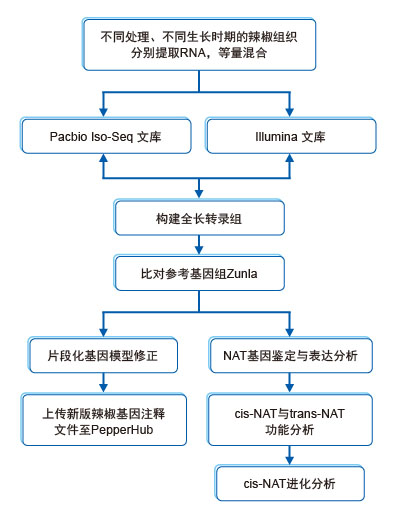
全长转录组测序揭示辣椒发育和逆境反应中广泛存在的天然反义转录基因
Full-length mRNA sequencing and gene expression profiling reveal broad involvement of natural antisense transcript gene pairs in pepper development and response to stresses
期刊:Plant Journal 发表时间:2019.04 影响因子:5.775 发表单位:华中农业大学、湖南省农科院
本项研究利用全长转录组测序提高了辣椒基因组的注释水平,为研究其功能基因组学提供了新思路;分析了NAT基因在辣椒生长过程中的广泛作用以及cis-NAT的进化机制。
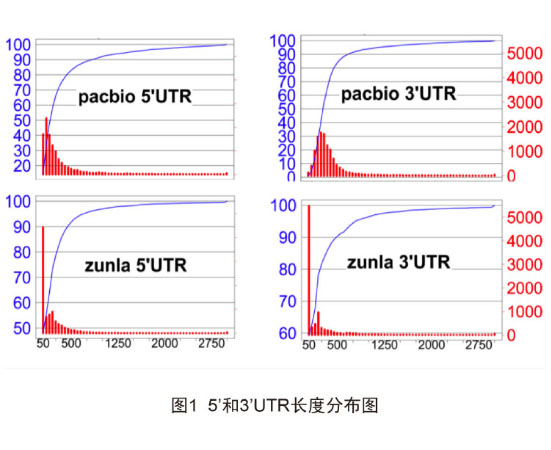
通过PacBio Iso-Seq测序,研究中共获得了57,962条高质量全长转录本,新发现基因5,769个。全长转录组测序的优势之一是可以获得mRNA的完整UTR区域,对与参考基因组拥有相同CDS区域的全长转录本进行分析,新测得的5'和3'UTR分别最高可达100nt和250nt,远远高于参考基因组的平均50nt(图1),这表示全长转录组测序可以捕获更多完整的基因结构。
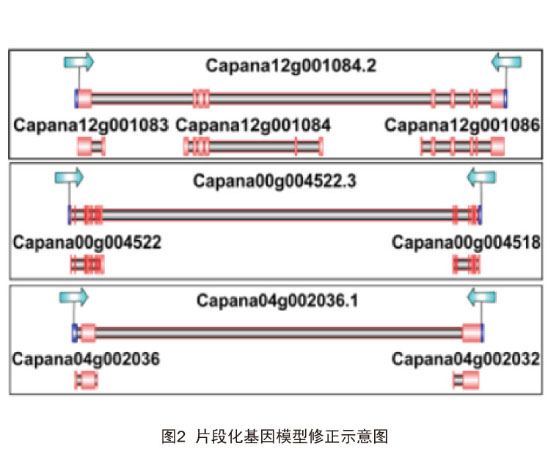
此前辣椒的基因注释主要基于短读长测序,对低表达基因外显子的覆盖度不够,导致一些基因模型的构建呈片段化。本次研究中利用PacBio测序修正了近500个注释错误,使片段化的基因模型趋于完整,被修正的模型多数含有较多的外显子或较长的内含子区域(图2)。
研究中通过全长转录组测序共获得2,057个cis-NAT和5,880个基因的34,193个trans-NAT,后者55.1%的互补序列都来源于转座子(图3)。通过分析NAT的表达模式,发现它们参与了辣椒的多种生物过程如辣椒素的合成等(图4);同时,对茄科植物进行了cis-NAT特点及进化分析,发现转座子可能参与新的cis-NAT形成(图5)。
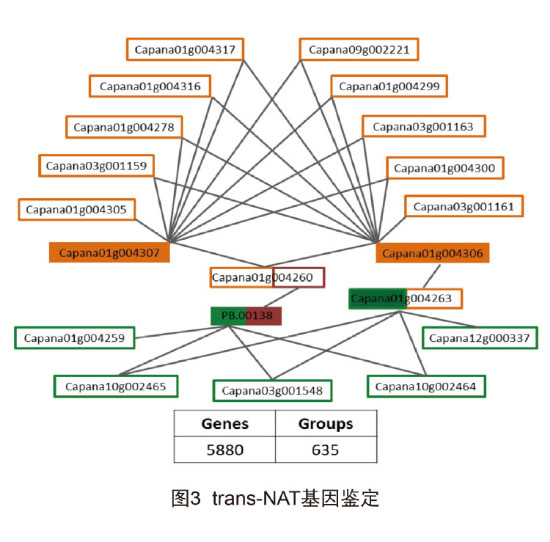
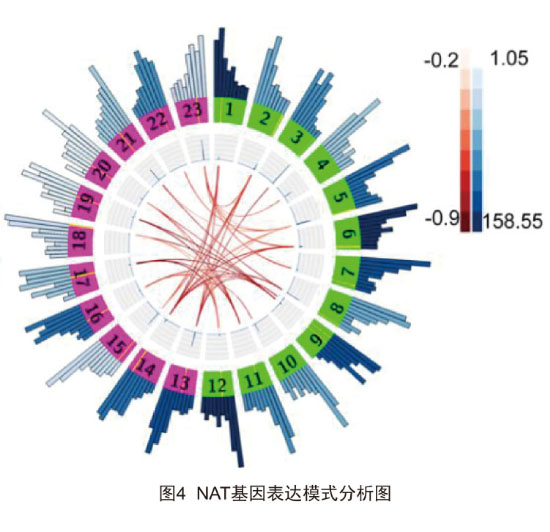

该研究利用全长转录组测序提高了辣椒基因组的注释水平,在基因组范围内分析了NAT基因的功能和进化过程,为研究辣椒的功能基因组学提供了新思路。
Wang J, Deng Y, Zhou Yi, Liu D, Yu H, Zhou Y, Lv J, Ou L, Li X, Ma Y, Dai X, Liu F*, Zou X*,Ouyang B*, Li F*. Full-length mRNA sequencing and gene expression profiling reveal broad involvement of natural antisense transcript gene pairs in pepper development and response to stresses. Plant J, 2019, 99: 763-783.