单体型基因组组装技术区别于传统嵌合基因组,其可以获得单独来自父本或母本、能同时遗传给下一代的一组序列信息。染色体水平的单体型基因组可以实现等位基因或同源染色体的组装。
“私人订制”服务
针对客户物种和材料准备情况,有针对性地定制测序和组装策略,定期反馈项目进展。
自主研发的分析流程
自主研发的分型流程,实现较高的准确度,较长的Block N50,较快的分型速度。
多种组装策略
可以实现有亲本二倍体、无亲本二倍体、异源/同源四倍体的有效分型。
经验丰富的分析团队
团队成员在Cell、Nature Communications等杂志发表过文章,项目经验丰富。
建立标准参考基因组
准确解析性状差异
等位基因特异性转录/表达研究
基因组编辑方案
单体型基因组组装揭示茶树的演化史
Haplotype-resolved genome assembly provides insights into evolutionary history of the tea plant Camellia sinensis
期刊:Nature Genetics 合作方式:提供服务 影响因子:38.330
发表时间:2021.07 合作单位:福建农林大学、中国农业科学院(深圳)农业基因组研究所 研究对象:茶树
茶叶作为一种全球性的经济作物,有很强的保健作用。茶树是无性繁殖的,这种方式可以有效地维持因有性重组而分离或丢失的有价值的基因型。然而,这种繁殖方式也会积累大量有害突变,导致“穆勒棘轮”效应,致使作物遭受损失。茶树是二倍体,含有15对同源染色体,嵌合式的基因组组装(筛选同源染色体中的一份拷贝作为代表组装到染色体水平)可能会错过重要选择性状的等位变异,而分型组装(不同亲本的两套同源染色体同时组装到染色体水平)能更完整地呈现二倍体基因组的全部遗传信息。
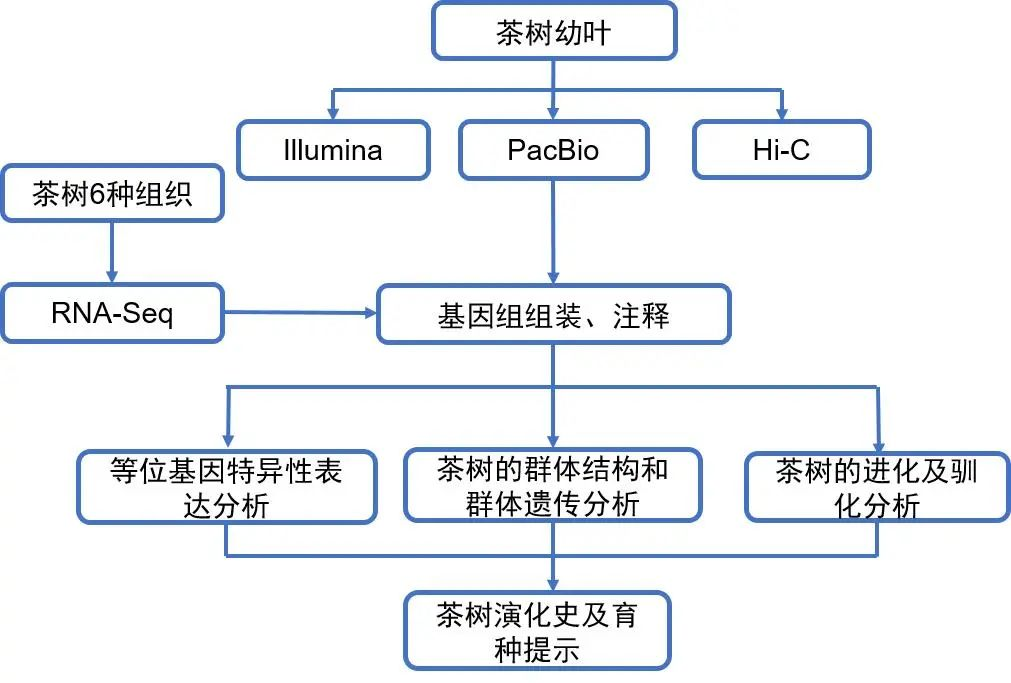
材料选择:山茶植株芽、根、茎、花、幼叶和成熟叶
测序策略:
DNA:
PacBio Sequel II平台基因组测序 114X
Illumina NovaSeq 150 bp双端测序,DNA小片段文库
Illumina NovaSeq,Hi-C文库 99.4X
RNA:
PacBio Sequel II平台,Iso-Seq文库
中国乌龙茶品种铁观音及几个主要的茶树品种和近缘物种进行测序及单体型组装,探索地理上不同的茶树群体之间的遗传多样性,为深入了解茶树的驯化史和进化史提供依据。
基因组组装与注释
铁观音的基因组大小约为3.15 Gb,杂合度为2.31%。利用PacBio长读长对原始数据进行组装得到初始contig,大小为5.41 Gb。将Khaper算法过滤产生的单倍体组装结果挂载到15个染色体(图1),得到单倍体参考基因组(monoploid reference genome),大小为3.03 Gb。同时利用ALLHiC算法得到铁观音单体型基因组(haplotype-resolved genome),大小为5.98 Gb。共线性分析显示它们的基因顺序高度一致。
 图1 铁观音基因组组装和质量评估
图1 铁观音基因组组装和质量评估
等位基因特异性表达
利用铁观音不同组织的全基因组测序,分离得到14,691个等位基因(图2),其中1,528个基因存在一致性的等位特异性表达(consistent allele-specific expression, ASEGs),即一个等位基因在所有组织和样本中的表达都高于另一等位基因。基因富集分析显示这些基因参与核糖体等多个生物学基本过程,与克服有害突变的潜在机制相关。同时还发现了386个非一致的ASEGs,它们在不同组织的等位基因之间存在特异性表达。其中几个基因与挥发性有机化合物的生物合成有关,包括黄酮和黄酮醇等的生物合成途径,这与植物的适应性演化相关。结果表明,在铁观音基因组中,一致性的ASEG明显多于不一致的ASEG(1,528
vs 386),这一趋势与杂交水稻的结果正好相反,即在杂交水稻中,不一致的ASEG远远大于一致性的ASEG。这种现象或许可以用杂种优势理论中的显性效应解释,长期无性繁殖的茶树利用优势等位基因应答不断积累的遗传负荷,以保持个体的适应度。
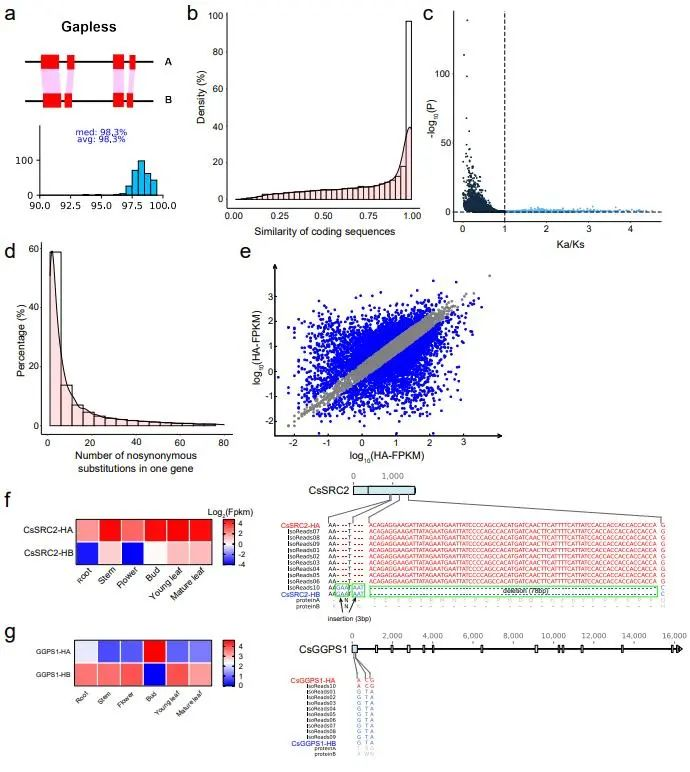
图2 茶树单倍型基因组等位不平衡
茶树遗传变异和群体结构分析
通过对161份茶树种质资源重测序数据分析,发现样本主要分为三类(图3),分别为大理茶,大叶茶和小叶茶,与茶树的形态学分类一致。另外大叶茶可以分类古大叶茶和栽培大叶茶;而小叶茶依据地理分布可分为四个亚组,分别为SSJ(陕西,四川,江西),ZJNFJ(浙江和福建北部),SFJ(福建南部),HHA(湖北,湖南和安徽)。TreeMix分析发现这些茶树之间存在显著的基因流动,表明种内基因交流频繁,其中一些与有记录的茶树杂交育种历史相吻合。
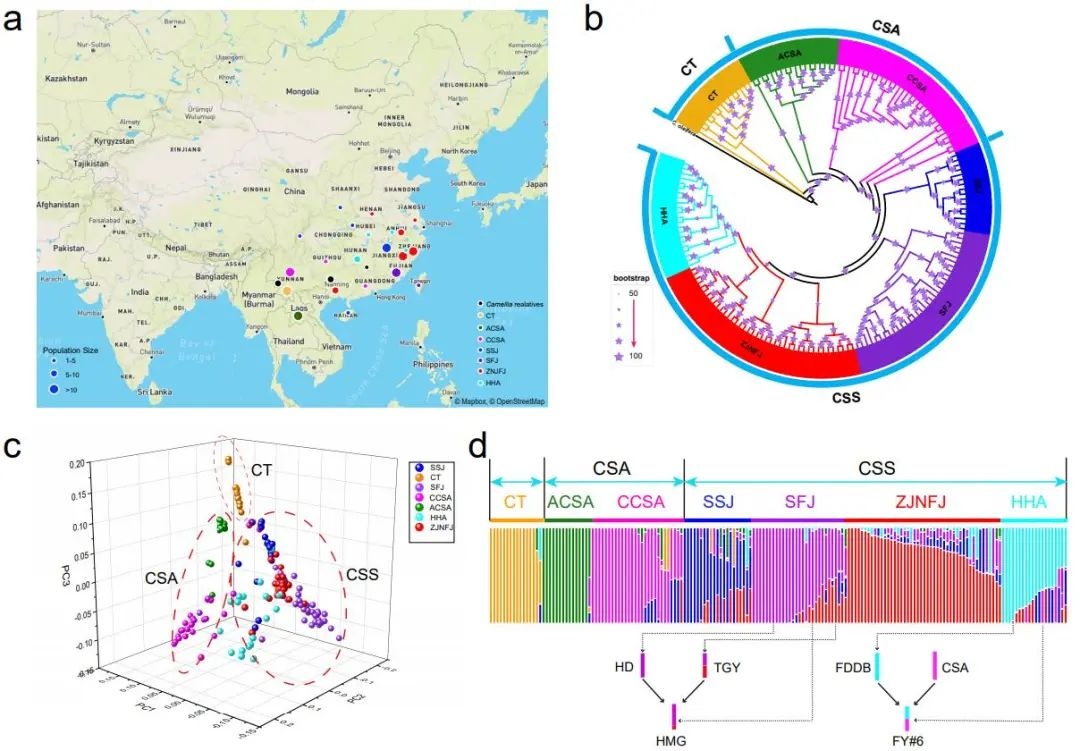 图3 茶树群体系统进化与群体结构分析
图3 茶树群体系统进化与群体结构分析
大叶茶和小叶茶的进化史和驯化史
对14种山茶属植物的21株单株进行了全基因组测序,通过群体遗传分析发现大叶茶和小叶茶具有不同的进化史。在Gelasian epoch时期(259-181万年前),剧烈的气候变化很可能导致了整个茶树物种(包括大叶茶和小叶茶)的群体收缩;两个变种分化后,仅小叶茶在Last Glacial Maximum时期(2.65-1.9万年前)可能由于温度骤降出现了再一次的群体收缩,但随后适应了环境的小叶茶迅速扩张,群体规模得到恢复。该分析表明,大叶茶和小叶茶分化后的进化史不同(图4)。通过对大叶茶和小叶茶驯化基因的分析,发现它们的驯化过程是并行的(即独立驯化),这些驯化基因参与了一系列重要的生物学过程且受人工选育的偏好性影响。基于KEGG分析,在大叶茶驯化早期以参与氧化石墨烯苷转运、糖苷转运通路为主,后期品种改良主要集中在合成生物碱和芳香化合物等。例如,研究人员鉴定到CsXDH基因在大叶茶品种改良阶段受到强烈的人工选择,该基因编码黄嘌呤脱氢酶,是咖啡因合成通路的重要基因。而小叶茶品种的早期驯化与植物抵御相关,改良过程主要集中在花发育的调控和对一氧化氮的响应,已有研究表明,NO的积累可以加速γ-氨基丁酸的消耗从而帮助植物抵御冷胁迫,这表明筛选耐寒的品种也是人工选育的重要目标。
 图4 大叶茶和小叶茶的平行驯化
图4 大叶茶和小叶茶的平行驯化
本研究成功组装了两套铁观音基因组(单倍体参考基因组和单体型基因组)。通过对等位基因特异性表达的分析,预测显性效应可能是铁观音应对遗传负荷的重要机制。通过对茶树种群水平的遗传分析,揭示了该物种的进化和人工驯化历史。该成果为利用组学分析和分子生物学技术挖掘功能基因、解析其背后的遗传调控机制,开展基于大数据驱动的基因组智能设计育种奠定了坚实的理论基础,同时也为缩短育种周期、提高育种效率、降低育种成本提供了科学依据。
Zhang X, Chen S, Shi L, et al. Haplotype-resolved genome assembly provides insights into evolutionary history of the tea plant Camellia sinensis[J]. Nature Genetics ,2021,53(8):1250-1259


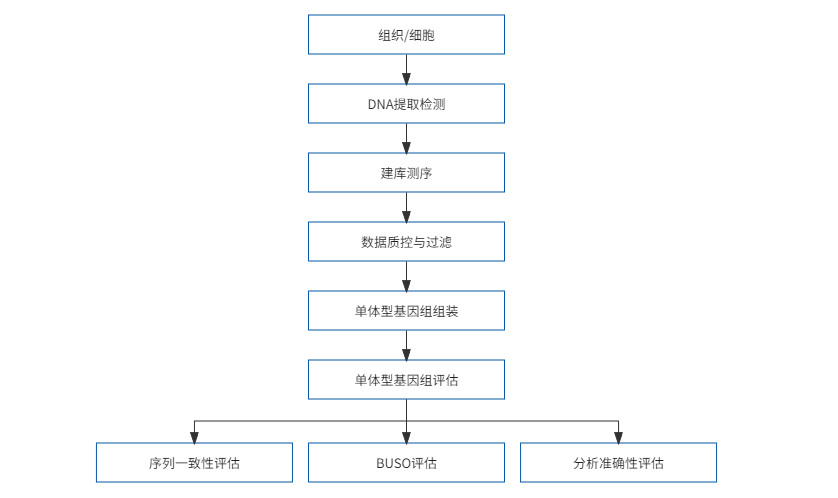

 图1 铁观音基因组组装和质量评估
图1 铁观音基因组组装和质量评估
 图3 茶树群体系统进化与群体结构分析
图3 茶树群体系统进化与群体结构分析 图4 大叶茶和小叶茶的平行驯化
图4 大叶茶和小叶茶的平行驯化