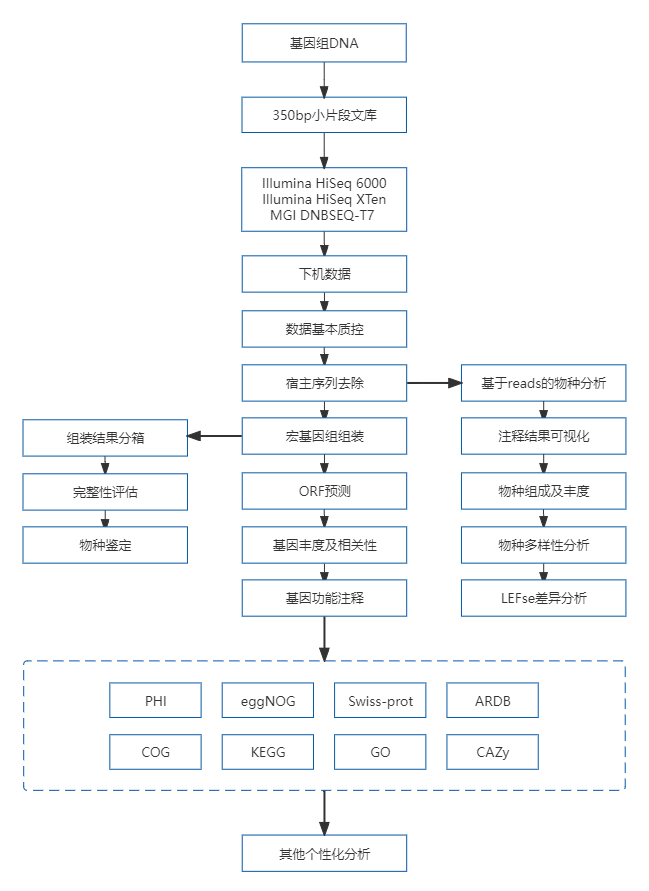
宏基因组是利用高通量测序技术以特定环境下微生物群体基因组为研究对象,在分析微生物多样性、种群结构、进化关系的基础上,进一步探究微生物群体功能活性、相互协作关系及与环境之间的关系,发掘潜在的生物学意义。
肺部宏基因组测序提示免疫功能低下儿童的漏诊感染
Pulmonary metagenomic sequencing suggests missed infections in immunocompromised children
期刊:Clinical Infectious Diseases 发表时间:2019 影响因子:9.079
发表单位:陈·扎克伯格生物中心,加州大学旧金山医学院,美国加州大学旧金山分校医学院生物化学与生物物理学系
目前尽管抗肿瘤和移植治疗的安全性有所提高,肺炎等感染性并发症的风险仍然很高。由于使用抗菌药物对培养物生长的抑制作用,血清免疫功能受损,以及多重检测预先选择的靶点有限,目前微生物学诊断往往无法识别病原体,导致大量死亡。因此,需建立一种高灵敏度的宏基因组测序技术来鉴定免疫功能低下儿童肺部病原体。
材料:34名具有肺部疾病且免疫功能低下儿童的41份下呼吸道样本
方法:样本经过机械均质化、提取RNA/DNA和宏基因组测序。将测序获得序列与NCBI核苷酸参考数据库进行比对以确定种属。根据每个样本中的丰度以及相对于其他样本的丰度来确定潜在致病微生物。
细菌:在检测队列的所有样本中,绝大多数来自细菌比对的分类单元丰度较低,并且数量相似。在<10 rpm且队列平均值的2个标准偏差内,鉴定出81.7%的细菌。包括潜在的致病细菌,它们在几乎所有样品中的水平都远高于阴性对照中检测到的水平。相反,只有0.4%被检测到的细菌符合异常标准。这些异常值包括在13/41个患者样本中鉴定出的10种潜在致病细菌。具有异常细菌病原体样本的细菌微生物组的α多样性明显降低。

图1 在免疫受损儿童的肺部检测到微生物

图2 具有异常病原体的呼吸道样本细菌α多样性降低
真菌:真菌的检出在该人群中普遍较少。92.1%的真菌定量在1 rpm以下。只有3.4%的真菌符合异常标准;包括从7/41个患者样本中被鉴定出的潜在致病真菌。虽然在14/41的患者样本中可检测到曲霉RNA,但通过培养和半乳甘露聚糖测定法,仅有1例浸润性肺曲霉病(IPA)阳性。尽管其余的13个都是培养阴性的,但其中12/13的样本在采集的48小时内已接受了抗曲霉药物治疗,这表明经验性的抗真菌药物治疗显著混淆了曲霉RNA与培养物生长之间的联系。曲霉RNA的水平与培养物生长无关,但与BAL半乳甘露聚糖试验呈弱相关。
病毒:与细菌和真菌相反,病毒的RNA比对呈现高丰度,并且符合异常标准的比例达到30.5%。在大约1/3的患者样本中发现了具有高丰度比对的传染性呼吸道病毒(13/41)。此外,有2例患者发生了病毒共感染。尽管没有临床上可疑的疱疹病毒性肺炎病例,但在9/41个样本中发现了低丰度的EB病毒、巨细胞病毒、HHV-6和HHV-7;在21/41个样本中还鉴定到致病性不确定或不太可能致病的病毒。
与临床试验的比较:临床检测在41.4%的样本中发现了致病病原体。其中,11个被mNGS一致地鉴定为离群值,这11个中有3个含有与第二个先前未检测到的潜在共病原相一致的离群值的RNA。另有3例经mNGS鉴定为离群值,另有3例经mNGS鉴定为不同的异常病原体。临床检测未在58.5%的样本中发现任何病原体。mNGS能够在统计上识别出24例病例中的11例外部潜在病原体。同时经过正交验证表明,mNGS在检测潜在肺部病原体方面比目前的临床诊断方法具有明显更高的敏感性。

图3 临床实验室结果与mNGS结果的比较
研究者提出了一种优化的mNGS检测方法,该方法揭示了免疫缺陷儿童中丰富的细菌、真菌和病毒性肺微生物组, 并在一半的临床阴性样品中鉴定出潜在的致病菌。因此, 先进的微生物检测为早期实施靶向性疗法提供了潜力,并为免疫受损儿童改善临床预后提供了可能性。
Zinter, S Matt, Dvorak, et al. Pulmonary Metagenomic Sequencing Suggests Missed Infections in Immunocompromised Children[J]. Clinical Infectious Diseases, 2019.