非编码区(Non-coding region)占人类基因组的绝大部分(约98%),它们不直接编码蛋白质,但在基因表达调控中起着重要作用。非编码区的结构变异,如平衡易位(Balanced translocation)、倒位(Inversion)等,可以影响染色质的三维结构和基因表达,从而引发肿瘤及遗传疾病等。正如生物学者常言:结构与功能相适应。因此,精度解析染色体的三维构象对于理解基因表达调控机制、揭示疾病发生发展的分子基础以及开发新的诊断和治疗策略具有重要意义。
近期,中山大学中山眼科中心张清炯教授团队联合中山大学中山医学院唐忠辉教授团队在Nature Communications(IF=14.7)发表了关于Altered chromatin topologies caused by balanced chromosomal translocation lead to central iris hypoplasia的研究成果,该研究通过深入地系统分析和功能验证,揭示了平衡染色体易位导致的染色质拓扑结构改变引发中央虹膜发育不全(Central iris hypoplasia, CIH)的分子机制,为相关领域的研究提供了新的视角和思路。
安诺优达有幸为该研究提供了Hi-C及RNA-seq分析服务。


样本材料:收集了同家系6例中央虹膜发育不全患者和6例正常对照的临床资料和外周血样本
测序方法:Hi-C互作、RNA-seq、WGS、WES、Sanger测序、长读长测序等

非编码区结构变异:研究发现,CIH的致病机制与非编码区的结构变异密切相关。这种变异通过改变染色质的三维结构,影响基因的表达调控。
染色质三维结构重建:平衡易位变异介导了染色质三维结构的重建,形成了新的TADs,使得异位增强子簇能够异常调节靶基因的表达。
基因表达异常:APCDD1基因在CIH患者中的表达显著增加,这与异位增强子的调控密切相关。APCDD1基因在虹膜成纤维细胞中的过表达进一步导致了虹膜发育不全的表型。

01 全基因组连锁分析:研究将CIH的致病基因定位在染色体6q15-q23.3和染色体18p11.31-q12.1两个区域。
02 全外显子组测序(WES)和全基因组测序(WGS)分析:排除了全部功能基因的编码区和非编码区变异。
03 长读长测序及连锁定位:发现了一个6号染色体和18号染色体的平衡位易变异t(6;18)(q22.31;p11.22)及其断裂点,并通过荧光原位杂交(FISH)和核型分析,证实了这种易位变异。
04 Sanger测序:证实在6个患者中存在变异断裂点,6个正常对照中不存在结构变异情况,家系中与疾病完全共分离,最终确定该复杂结构变异是CIH的病因。
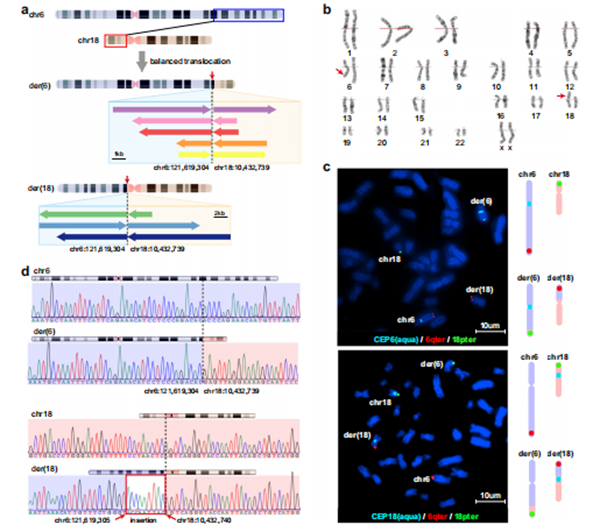
图1 鉴定非编码区平衡易位变异
05 Hi-C分析:研究团队利用Hi-C技术对来自患者及其正常同胞的诱导性多潜能干细胞(iPSCs)进行了全基因组染色质三维结构检测,发现患者样本的iPSCs在平衡易位断点附近形成了新的拓扑关联结构域(TADs),这些新的TADs使得18号染色体上的APCDD1与6号染色体上的增强子之间的异位染色质相互作用,从而导致APCDD1在患者中显著上调。
06 RNA-seq分析:发现断点附近的众多基因中,仅APCDD1基因的表达在病人中显著增加。这提示结构变异形成的新的TAD使得异位增强子簇异常调节该TAD中的靶基因APCDD1,导致其表达上调。
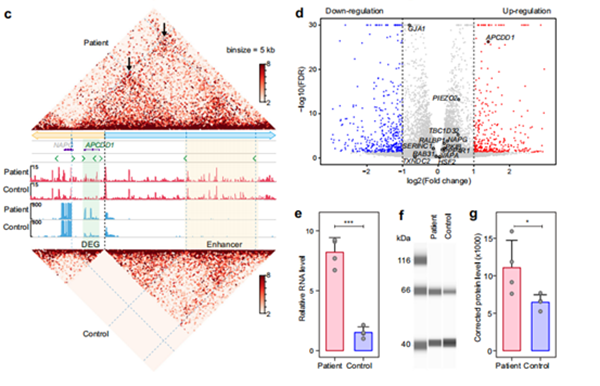
图2 非编码区结构变异引起的染色质三维结构和基因表达改变
07 敲除实验和斑马鱼模型验证:进一步证实APCDD1基因的过表达与异位增强子的调控相关,且APCDD1基因过表达与眼球发育缺陷的关联。

本项研究揭示了非编码区域的结构变异如何通过改变染色质空间构象,进而影响基因表达,最终导致单基因遗传病的发生。研究强调了非编码区域在调控基因表达和单基因病发生中的重要作用。为开拓特殊非编码区结构变异导致单基因遗传病这一新领域具有重要意义。为未来针对CIH及其他类似遗传病的诊断和治疗提供了新的思路和方法。

低起始量Hi-C技术---降低Hi-C技术建库“门槛”
传统的Hi-C技术由于其需要大规模的细胞起始量,在应用于生殖生物学、肿瘤学以及其他涉及微量样本的研究领域时面临着巨大挑战。因此,降低Hi-C建库起始量,一直是三维基因组学研究者想要攻关的难题。
安诺优达已成功推出了10万个细胞水平的Hi-C建库,实现了低起始量Hi-C建库的产业化服务,让Hi-C技术应用到了更多的研究领域。
对1个样本的2个文库进行小数据评估,结果显示,最终可用于分析的Valid Rate(%)均在90%以上,可用于大数据分析。
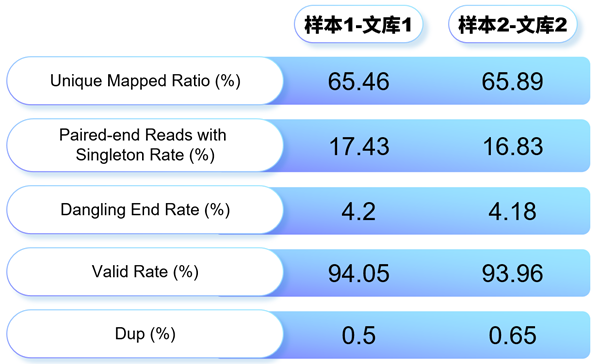
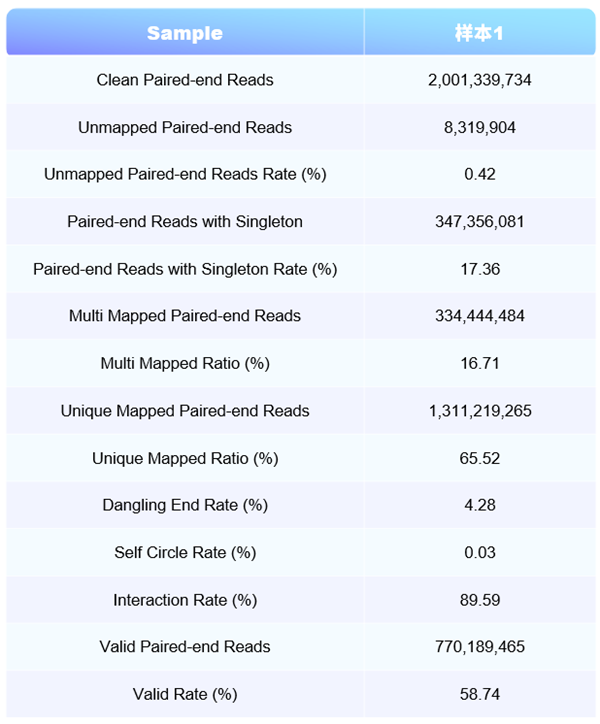
大数据上机,有效数据统计如下:
若您需要了解更多产品信息,欢迎通过安诺优达官网、微信公众号、当地销售或ts_support@genome.cn的邮箱等渠道与我们取得联系,期待为您服务。

